ИНСТИТУТ ЦИТОЛОГИИ РОССИЙСКОЙ АКАДЕМИИ НАУК
| Группа некодирующей ДНК |
|
Руководитель группы ПОДГОРНАЯ Ольга ИгоревнаД.б.н., проф., г.н.с.e-mail: opodg@yahoo.com |

|

к.б.н. Н.И.Енукашвили |

к.б.н. А.П.Воронин |

к.б.н. И.С.Кузнецова |

к.б.н. И.В.Матвеев |

А.Комиссаров |

Е.Гаврилова |

Н.Галактионов |

Л.Адонин |
|
ТЕКУЩИЕ ТЕМЫ 1. Классификация тандемных повторов (ТП) в базах данных генома мыши (in silico, биоинформатика) и проверка полученных предсказаний о позициях ТП in situ с помощью флюоресцентной гибридизации in situ (FISH). Найдены хромосом специфичные варианты ТП. 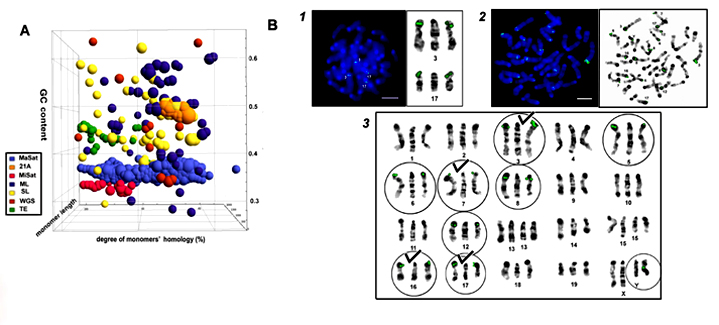
В WGS мыши идентифицировано около 200 групп предсказанных in silico тандемных повторов (ТП). Структуры типа HOR (повторы высшего порядка) найдены для многих ТП, что предполагает наличие их хромосом-специфичных вариантов. Пробы для трех представителей семейств сконструированы на основе мономеров. Все хромосомы, несущие ТП in silico, метятся и in situ, однако метятся и другие хромосомы из-за неполной сборки гетерохроматиновых районов в эталонном геноме. Длинная проба, основанная на варианте TRPC-21A с хромосомы 3, гибридизуется на концах хромосом 3 и 17. До сих пор не существовало хромосом-специфичных проб на основе тандемных повторов для цитогенетики мыши. Используя полученные данные, предполагается картировать часть новых ТП, имея в виду найти хромосом-специфичное распределение. Хромосом-специфичность проб может быть усилена мультимеризацией, так как длинная проба оказалась успешной. Пробы будут усовершенствованы вплоть до коммерческого использования с помощью компании Бигль (Санкт-Петербург). 2. Недавно открыта транскрипция сателлитной ДНК (сатДНК, разновидность тандемных повторов). Мы показали, что сатДНК вовлечена в ассоциацию хромосомных территорий. Проникающая на соседнюю хромосомную территорию сатДНК до некоторой степени деконденсируется. Деконденсация сопровождается деметилированием и транскрипцией. При дифференцировке мышиных эмбриональных стволовых клеток E-14 и IOUD2 перицентромерная сатДНК попадает в хромоцентры только через несколько дней после начала индукции. Перемещение сатДНК сопровождается ее ассоциацией с белком гетерохроматина HP1a и транскрипцией с перицентромерных, но не центромерных, тандемных повторов (ТП). РНК транскрибируется с одной (прямой) цепи и полиаденилирована. Пробы, полученные с этой РНК как кДНК, гибридизуются со всеми хромосомами. В дифференцированных клетках транскрипты находятся исключительно в хромоцентрах, в стволовых же и в клетках в процессе дифференцировки транскрипты формируют 1-2 кластера вне хромоцентров. Используя хроматин иммуно-преципитацию (ChIP) и иммуно окрашивание, мы показали, что в индуцированных клетках перицентромерные ТП (сатДНК) взаимодействуют с р68 РНК-хеликазой, которая известна как белок, вовлеченный в регуляцию транскрипции и дифференцировку. 3. Нашли новый белок, мезоглеин, и клонировали его ген. Тело сцифоидной медузы Aurelia aurita состоит из 2 слоев - эпидермиса и гастродермы. Слои разделены толстой прослойкой внеклеточного матрикса - мезоглеей. У A. aurita мезоглея населена мезоглеальными клетками. Мажорный белок мезоглеи с мол.массай 47 кДа определили с помощью диск-электрофореза в денатурирующих условиях (SDS-PAGE). Клонировали и сиквенировали часть мРНК этого белка длиной 1421 н.п. Поиск гомологии по нуклеотидным и белковым последовательностям показал, что это новый белок. Компьютерно полученная белковая последовательность содержит 416 аа домен ZP (Zona pellucida) и домен Delta/Serrate/Lag-2 (DSL). Белок назвали мезоглеин. Мезоглеин экспрессируется только в мезоглеальных клетках взрослой медузы, как показал ПЦР на РНК из разных типов клеток. Мезоглеин является представителем ZP доменных белков для самого низшего из типов животных. Белок оказался не только структурным элементом мезоглеи медузы, но и входит в состав новой структуры - пластины контакта в ооците A. aurita. 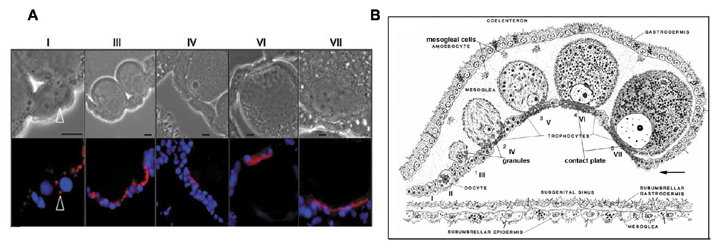
4. Иглокожие - вторичноротые животные, которые рано отделились от ветви, ведущей к млекопитающим. Положение на филогенетическом дереве, доступность и свободное развитие в морской воде, сделало иглокожих, особенно морских ежей, модельными объектами для решения многих ключевых вопросов биологии развития, биохимии, клеточной и молекулярной биологии, эволюционной биологии (Kondo et al., 2012). Морские ежи семейства Strongylocentrotidae особенно популярны. Их обилие, большой размер и легкость содержания в лабораторных условиях, привели к тому, что они стали объектом многих ранних работ по биологии развития (Gustafson and Wolpert, 1964; Monroy and Maggio, 1964; Neyfakh, 1971). В 2006 году опубликован геном Strongylocentrotus purpuratus, известного как "пурпурный морской еж" (Sea Urchin Genome Sequencing Consortium). Это один из немногих хорошо аннотированных геномов иглокожих. Мы воспользовались преимуществами прочитанного генома Strongylocentrotus purpuratus для того, чтобы выявить транспозоны (TE, Transposable Element) и проверить наличие некоторых из них в геноме S. intermedius - дальневосточного серого морского ежа. Работа представляет собой необходимый подготовительный этап, чтобы использовать подобранные и проверенные праймеры для изучения транскрипции соответствующих ТЕ в развитии серого морского ежа. Расхождение видов морских ежей произошло около 10 млн лет назад (Youn-Ho Lee, 2000). Мы использовали известные TE S. purpuratus, содержащиеся в базе данных повторов RepBase для расчета праймеров и выявления соответствующих ТЕ в транскриптоме S. intermedius. С помощью геномного ассемблера SPADES (с параметрами по умолчанию) собрали из ридов транскриптома серого морского ежа, картированных на соответствующий мобильный элемент пурпурного морского ежа последовательности первых (по представленности) 100 мобильных элементов (см.: Табл.1). В таблице также представлены подобранные к последовательностям ТЕ праймеры и результаты ПЦР-анализа. При ПЦР анализе мы получили продукт расчетной длинны в 92 случаях (из 100). Таким образом, результаты работы подтверждают оправданность использованного подхода. Экспериментальная работа с праймерами, сконструированными на основе ТЕ пурпурного ежа для идентификации ТЕ серого морского ежа, привела бы к неверным результатам: отсутствие транскрипции было бы артефактно основано на несходстве последовательностей. Проверенные и давшие положительный результат 92 из 100 собранных последовательностей ТЕ серого морского ежа безусловно пригодны для дальнейшей работы. Восемь пар праймеров, которые не дали ампликона расчетной массы или же дали набор продуктов (шмер), рассчитаны для последовательностей, количество ридов которых в транскриптоме невелико, что повлекло за собой ошибки сборки. Мы надеемся исправить эти ошибки после сборки прочитанного генома серого морского ежа. Работа группы поддержана грантами РФФИ и грантом Президиума Академии наук "Молекулярная и клеточная биология" |
|
Komissarov AS, Gavrilova EV, Demin SJ, Ishov AM, Podgornaya OI. 2011. Tandemly repeated DNA families in the mouse genome. BMC Genomics. 12(1):531. (http://www.biomedcentral.com/1471-2164/12/531) Matveev I.V., Adonin L.S., Shaposhnikova T.G. Podgornaya O.I. 2011. Aurelia aurita - Cnidarian with a prominent medusiod stage. J Exp Zool B Mol Dev Evol. 2011 Nov 11. doi: 10.1002/jez.b.21440. [Epub ahead of print] А.С. Комиссаров, И.С. Кузнецова, О.И. Подгорная. 2010. Центромерные тандемные повторы мыши in silico и in situ. Генетика, 46, № 9, 1217-1221 О.И. Подгорная, Д.С. Остромышенский, И.С. Кузнецова, И.В. Матвеев, А.С. Комиссаров. 2009. Парадоксы организации центомера и гетерохроматина. Цитология, 51 (3): 204-11. Остромышенский Д.И., Кузнецова И.С., Картавцева И.Н., Голенищев Ф.Н., Маликов В.Г. Подгорная О.И. Сателлитная ДНК как филогенетический маркер и границы его применимости на примере трех родов Murinae. Цитология, в печати Подгорная О.И., Галактионов Н.К. (2009) Мобильные элементы как потенциальные векторы горизонтального переноса генетической информации в системах паразит-хозяин. Труды зоологического института РАН. 313 (3); 283 - 296 И.С. Кузнецова, Е.М. Нониашвили, Е.В. Гаврилова, А.П. Дыбан (2009) Изменение метилирования мажорного сателлита в ядрах двухклеточных мышиных зародышей в зависимости от условий развития. Онтогенез, 40, 5: 379-387 Адонин Л.С., Подгорная О.И., Матвеев И.В., Шапошникова Т.Г. (2009) Пластинка в зоне контакта ооцита с зачатковым эпителием у сцифомедузы Aurelia aurita имеет иммунологическое сходство с ZP-домен-содержащим белком мезоглеином. Цитология, 51 (5): 435-441 Адонин Л.С., Подгорная О.И., Шапошникова Т.Г. Морфологическая структура пластинки в зоне контакта ооцита с зачатковым эпителием у сцифоидной медузы Aurelia aurita (Cnidaria: Semaeostomae) // Онтогенез. М., в печати. Kukalev A.S., Lobov I.B, Percipalle P., Podgornaya O.I. 2009. SAF-A/hnRNP-U localization in interphase and metaphase. Cytogenet Genome Res. 124 : 288-297. Enukashvily N. I., Malashicheva A. B., Waisertreiger I. S-R. 2009. Satellite DNA spatial localisation and transcriptional activity in mouse embryonic E-14 and IOUD2 stem cells. Cytogenet Genome Res. 124 : 277-287. Enukashvily N. I., Donev R., Waisertreiger I. S., Podgornaya O. I. 2007. Human chromosome 1 satellite 3 DNA is decondensed, demethylated and transcribed in senescent cells and in A431 epithelial carcinoma cells. Cytogenet Genome Res. 118 (1) : 42-54. Matveev I.V., Shaposhnikova T.G, Podgornaya O.I. 2007. A novel Aurelia aurita protein mesoglein contains DSL and ZP domains. Gene. 399 : 20-25. Kuznetsova I, Podgornaya O, Ferguson-Smith M.A. 2006. High-resolution organization of mouse centromeric and pericentromeric DNA. Cytogenet Genome Res. 112 : 248-255. Kuznetsova I.S, Voronin A.P, Podgornaya O.I. 2006. Telomere and TRF2/MTBP localization in respect to satellite DNA during cell cycle of the mouse cell line. Rejuvenation Research. 9 (3) : 391-401. Shaposhnikova T., Matveev I, Napara T, Podgornaya O. 2005. Mesogleal cells of the jellyfish Aurelia aurita produce the fibres of the mesoglea. Cell Biol Int. 29 : 952-958. Kuznetsova I.S., Prusov A.N., Enukashvily N.I., Podgornaya O.I. 2005. New types of mouse centromeric satellite DNAs. Chrom Res. 12 : 9-25. Enukashvily N., Donev R., Sheer D., Podgornaya O. 2005. Satellite DNA binding and cellular localisation of RNA helicase p68. J Cell Sci. 118 : 611-622. Podgornaya O.I., Voronin A.P., Enukashvily N.I., Matveev I.V., Lobov I.B. 2003. Structure-specific DNA-binding proteins as the foundation for 3-dimensional chromatin organization. Int Rev Cytol. 224 : 227-296. |